自然免疫応答の遺伝的多様性を単一細胞分解能で理解する
自然免疫応答の遺伝的多様性を単一細胞分解能で理解する
熊坂夏彦
東京大学医科学研究所 ヒトゲノム解析センター デジタル・ゲノミクス分野
Mapping interindividual dynamics of innate immune response at single-cell resolution
Natsuhiko Kumasaka, Raghd Rostom, Ni Huang, Krzysztof Polanski, Kerstin B Meyer, Sharad Patel, Rachel Boyd, Celine Gomez, Sam N Barnett, Nikolaos I Panousis, Jeremy Schwartzentruber, Maya Ghoussaini, Paul A Lyons, Fernando J Calero-Nieto, Berthold Göttgens, Josephine L Barnes, Kaylee B Worlock, Masahiro Yoshida, Marko Z Nikolić, Emily Stephenson, Gary Reynolds, Muzlifah Haniffa, John C Marioni, Oliver Stegle, Tzachi Hagai, Sarah A Teichmann
Nat Genet. 2023;55:1066-1075.
論文のハイライト
本研究の最も革新的な点は、世界に先駆けて自然免疫応答の転写レベルにおける非線形で連続的な遺伝的多様性を、一細胞分解能で示したことにある。そのためのin vitroシステムとして、68人の健常者(HipSciドナー)から得られた線維芽細胞株を用いて、ウイルス感染化での自然免疫応答を模倣するPoly(I:C)とInterferon-β(IFN-β)という2つの細胞刺激によって免疫反応の活性化をおこなった。その後、SmartSeq2と呼ばれるプレート単位の一細胞RNA解析技術を用いて、一細胞ごとの遺伝子発現量を定量化した。一細胞RNA解析の利点は、従来のバルクRNA解析では不可能であった細胞状態を連続的に捉えることができる点にある。一方で、対象となる細胞の状態以外にも細胞周期やドナー、実験バッチなど様々な内的・外的な交絡因子の影響を受けてしまうという欠点があり、データ解析には細心の注意が必要となる。
そこで我々は、一細胞データからターゲットとなる自然免疫応答に関わる潜在変数だけを抽出し、頑健な動的eQTL(expression quantitative trait locus)解析を可能にするGASPACHO (GAuSsian Processes for Association mapping leveraging Cell HeterOgeneity)という統計解析手法を新たに開発した。これは、単に遺伝子発現量だけを解析するのではなく、ドナーや実験プレートといった様々な既知の交絡因子と遺伝子多型データを入力データとして、ガウス過程潜在変数モデルによって頑健に細胞状態を推定し、且つ、ガウス過程回帰モデルによって非線形で動的なeQTLを網羅的に解析するという統合解析手法である(図)。
我々はこの手法によって線維芽細胞で発現しているゲノムワイド10,748遺伝子のなかで、1,275遺伝子のeQTLを同定した。さらに、この中で27%が自然免疫反応によって誘導される動的eQTLであることを明らかにした。これらの多くは免疫応答による発現量の上昇に伴って、関連の強さもまた連続的に上昇していくような特異的eQTLであり、ナイーブな細胞状態を解析しただけでは検出されないことも明らかになった。
次に、我々はOpenTargets(https://www.opentargets.org/)で収集された701のGWASの結果と、我々の同定したeQTLを共局在化解析し、823遺伝子に対する495の異なるGWAS形質における3,132ものeQTL―疾患関連座位ペアの共局在化を同定した。これらの共局在化は、免疫に関連するGWAS形質に偏っている事が明らかになり、自然免疫反応を発現レベルで変化させる変異の多くがGWASで同定された関連座位において機能的であることが示唆された。
一例として、COVID-19のGWAS関連座位であるOAS座位において、OAS1遺伝子の動的eQTLが強く共局在化していた(共局在化事後確率=0.99)。この領域には複数の多型がCOVID-19との関連を示しており、rs10774671が最も強い関連を示していた。このSNPはOAS1 eQTLにおいて最も強い相関を示した多型の一つであり、OAS1遺伝子の最終エクソンの5’側に存在する。OAS1は自然免疫の二次反応に呼応して発現が高くなっているが、GASPACHOによる動的eQTL効果の推定量からは、COVID-19のリスクアレルAが免疫応答下でOAS1遺伝子の発現を有意に低下させていることが明らかになった。
そこで我々はさらにrs10774671の機能を調べた結果、変異アレルAがイントロン終端に存在するCAGモチーフをCAAに変える事で、通常発現しているisoformとは異なる3つのisoformを生み出している事が明らかになった。これらのisoformはどれもOAS1のタンパク質発現異常に影響していることが示唆されており、また西ナイルウイルスの感染を上昇させることが報告されている。したがって、新型コロナウイルスに関しても高い確度で感染のリスク因子になっていることが明らかになった。
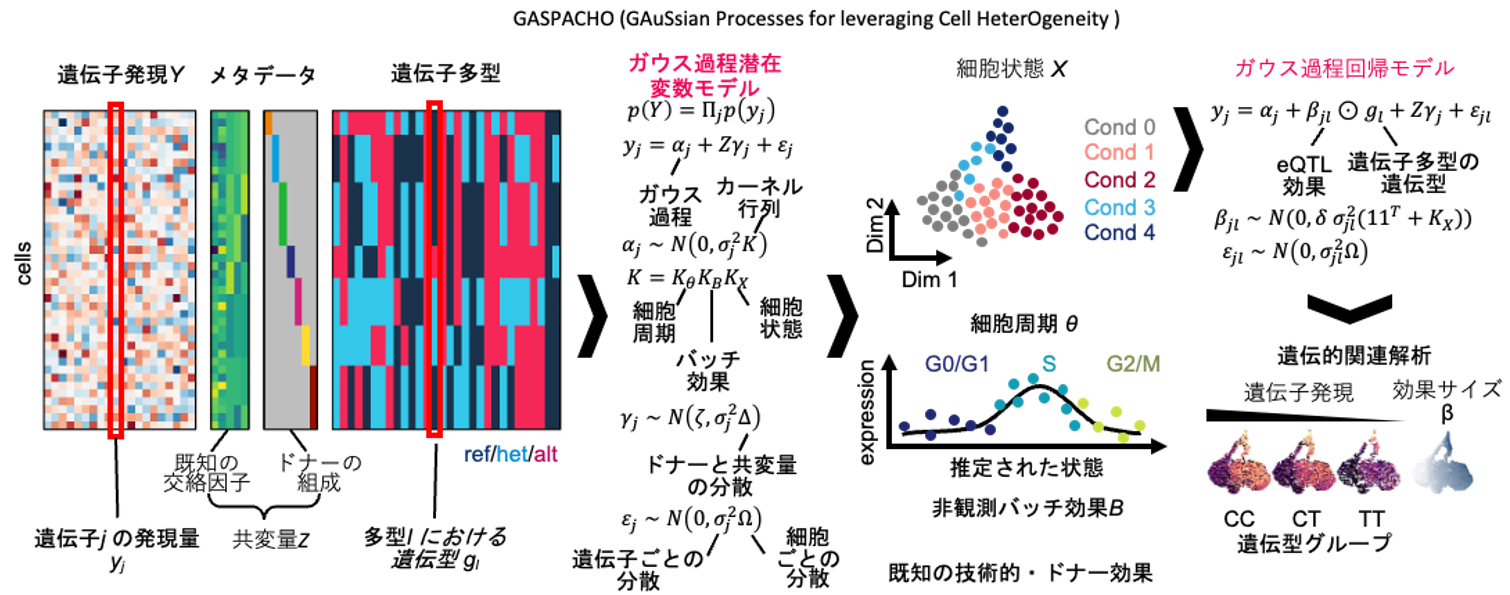
図:GASPACHO(GAuSsian Processes for Association mapping leveraging Cell HeterOgeneity)のワークフロー
工夫した点、楽しかった点、苦労した点など
本研究で扱ったヒト線維芽細胞の一細胞RNA解析データは、ケミストリーの変更によるバッチ効果など技術的なバイアスの影響が非常に大きなデータで(原著論文参照)、複数の研究者がデータ解析において匙を投げ、論文化が暗礁に乗り上げていたデータでした。しかしガウス過程潜在変数モデルという最新の統計解析技術を用いて、粘り強くデータの特徴を一から数理モデルとして記述する事で、免疫応答に関わる遺伝子においてCOVID-19をはじめ疾患と関連するようなeQTLが次々と同定されました。単に既存の手法を適用するだけでは一見してネガティブなデータであっても、すぐにあきらめて再実験を繰り返すのではなく、今手元にあるデータとじっくりと向き合ってデータの声に耳を傾けることが必要と再確認する良い経験でした。一研究者として独立してから今年で16年になりますが、今でも大学院時代の指導教授である柴田里程先生から与えられた2つの金言「データは現象の放つ光である」と「本質は細部に宿る」は私の研究活動におけるモットーとなっています。
研究室紹介
東京大学医科学研究所のデジタル・ゲノミクス分野(熊坂夏彦研究室)は2023年10月に設立された新しい分野として、最新の統計・機械学習技術と分子生物学的技術を組み合わせ、さらに医科研のスパコンSHIROKANEを駆使して、ゲノム上の特に非コード領域に存在するありふれた変異の分子機序を明らかにする事を目標に研究活動を行っています。まだ研究室のメンバーは私だけですが、今後、大学院生や研究員・教員が増え、活発で実り多い研究分野にしていきたいと考えています。

